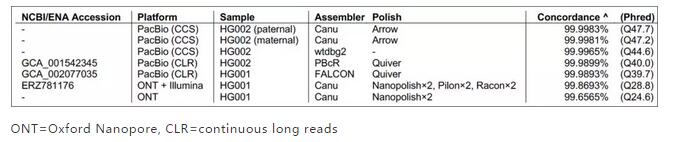
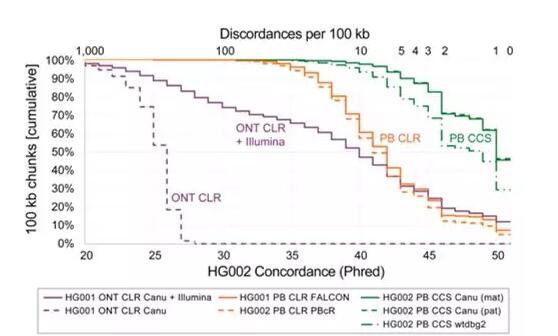
如今,人类基因组的测序已经达到群体规模,但仍需要结合不同的测序技术(短读长和长读长),才能覆盖各种类型的遗传变异。这无疑增加了测序项目的成本和复杂度。为此,Pacific Biosciences公司的研究人员开发出一种方案,能够在Sequel测序平台上产生高度准确的长reads。 在PacBio、Google、NIST等机构的共同努力下,研究人员对人类男性HG002样本进行了测序,覆盖度达28倍,平均读长为13.5 kb,且准确性高达99.8%。这项成果于本周发表在预印本网站bioRxiv上。PacBio公司的David Rank和Michael Hunkapiller为共同通讯作者。 实验结果 我们先来看一下实验结果。研究小组最终使用PacBio SMRT测序得到了高质量的数据,平均CCS subreads达到13.5kb,单条reads的准确性达到99.8%,总共获得了89 Gb CCS数据。有了这样高质量的测序数据做基础,de novo组装结果自然也非常惊艳,其contig N50达到了15Mb,与HG002/NA24385基因组吻合率达到99.998%(Q47.7)。这一组装结果无论是对比单独Nanopore测序组装方案(Q24.6)或Nanopore测序+Illumina测序组装方案(Q28.8)都具有明显优势。 从下图可以看出使用PacBio CCS reads组装出的contig质量达到Q50的有60%左右。而单独使用Nanopore reads组装的HG001基因组 contig 质量达到Q50 的是0%(没有任何contig 能达到Q50)。 即使使用Nanopore + Illumina 纠错后混装方案达到 Q50 的contig 也只有15%。 PB=PacBio, ONT=Oxford Nanopore, CLR=continuous long reads Contig chunk 在质量分数上的累积分布曲线 将PacBio CCS reads mapping到人参考基因组GRCh37进行比对,检测结构变异、SNV和Indel的准确率和召回率(Precision & Recall),均分别大于95.99%、99.91%及95.98%。并且99.64%检测出来的变异都进行了单倍体分型。检测结构变异本来就是PacBio的优势,通过CCS的方式,PacBio SMRT测序对SNV和Indel的检测能力已和Illumina测序技术相当。 总结:长读长优势何在 PacBio长读长提升复杂基因组组装指标 PacBio超长读长直接获得全长转录组信息 PacBio直接读取原始数据中DNA甲基化修饰信息 PacBio长读长组装还可获得完整的细胞器基因组 2013年,天津生物芯片率先在国内提供PacBio RS II三代测序服务,天津生物芯片已积累丰富的项目经验,成为中国三代测序技术应用的领跑者。截止目前,公司在Nature、Nature Communications、PNAS等杂志累计发表SCI论文超过130篇,总影响因子超过500。同时,于2016年签约引进了PacBio Sequel测序平台,为大家带来高性价比测序服务,并为各位合作伙伴提供大基因组全套解决方案。 PacBio SMRT测序目前已经在单reads准确率和低样本起始量(100ng DNA起始量文库)上取得了突破,而这两个优势搭配即将到来的8M测序芯片,未来必然会大大拓展SMRT测序的应用领域。

使用PacBio 13.5kb CCS文库组装高精度人类基因组结果发布
作者:天津生物芯片技术有限责任公司 2019-01-18T00:00 (访问量:4169)
天津生物芯片技术有限责任公司 商家主页
地 址: 天津经济技术开发区宏达街23号
联系人: 王小姐
电 话: 022-66229515
传 真: 022-66226985
Email:tjbiochipmk@126.com
相关咨询
开学献礼,天津生物芯片助力科研再启航 (2020-08-13T14:57 浏览数:1356)
PacBio技术助力两种水生生物的基因组组装 (2019-02-22T16:11 浏览数:3941)
gcMeta平台:有效支撑我国微生物组研究 (2018-11-02T09:52 浏览数:6513)
一伍一拾 ,TBC十五周年 回归本心 倾情回馈微生物学研究 (2018-06-01T11:42 浏览数:3888)
海洋微生物那么复杂, 天津生物芯片助力破解神之谜团 (2018-04-17T15:42 浏览数:3242)
天津生物芯片项目文章 |海参基因组并在形态进化与器官再生研究方面获重要进展 (2017-10-26T10:28 浏览数:3255)
TBC林木计划,夏不为利—“林木基因组”与“Pacbio三代测序”的完美碰撞 (2017-08-03T10:25 浏览数:3247)
当海洋生物基因组科研遇上三代测序--革首南极鱼(南极抗冻鱼) (2017-07-18T14:53 浏览数:5006)
天津生物芯片百万基金助力中国海洋水产生物基因组研究,等你来约! (2017-06-20T15:23 浏览数:2132)
科学家用SMRT测序探索极端微生物基因组 (2017-03-03T09:46 浏览数:3013)
